基因的差异表达造成了细胞分化,使得每个组织、器官在拥有相同基因基础上表现出了不同的形态和功能。这种差异表达也为植物针对外界环境信号做出响应提供了可行性。转录因子(transcription factors, TFs)也称反式作用因子(trans-acting factors)就是实现基因时空差异性表达的关键元素。转录因子能够识别并结合DNA上的顺式作用元件(cis-acting elements/cis-regulatory DNA sequences),进而调控相关基因的表达【1】。
植物体内的全部转录因子与其靶标基因共同构成一个基因调控网络(gene regulatory network, GRN),也就是一个蛋白与DNA的互作关系网。有一些研究通过高通量的方法【2】,比如ChIP-seq等,系统地描述了模式植物拟南芥在一些特定发育时期和特定环境条件下的GRN【3】,但是这些研究都只基于蛋白与DNA的物理互作,并没有深入其功能研究,也就是说,这些蛋白与DNA的互作是否真的对相关基因的表达具有调控作用并不清楚。
近日,来自比利时根特大学的研究团队在Nature Plants上发表了题为Integrative inference of transcriptional networks in Arabidopsis yields novel ROS signaling regulators的研究论文,建立了一个集成的基因调控网络(integrated gene regulatory network, iGRN)。该集成网络准确描述了681个转录因子的已知功能,并预测了数百个转录因子的新功能。
该研究通过监督式学习方法,输入了拟南芥中7个关于顺式作用元件、染色质、转录因子、基于表达调控的互作关系等数据库,生成了一个基因调控集成网络(integrated gene regulatory network, iGRN),该网络覆盖了1491个转录因子与31393个相关靶标基因的1709871个互作关系。通过与基于实验的数据分析(Y1H, TF ChIP–seq 以及 DE)的对比,研究人员证实了iGRN是一个可以被实验数据支撑的功能性互作网络。比如,iGRN预测的互作关系,有39%与在体内转录因子结合的TF ChIP–seq数据重叠。进一步的分析发现,iGRN数据中的转录因子与其靶基因的植物表型具有高度相关性,说明iGRN中体现的互作关系具有实际功能,即这些互作关系确实能够影响相关基因的表达。
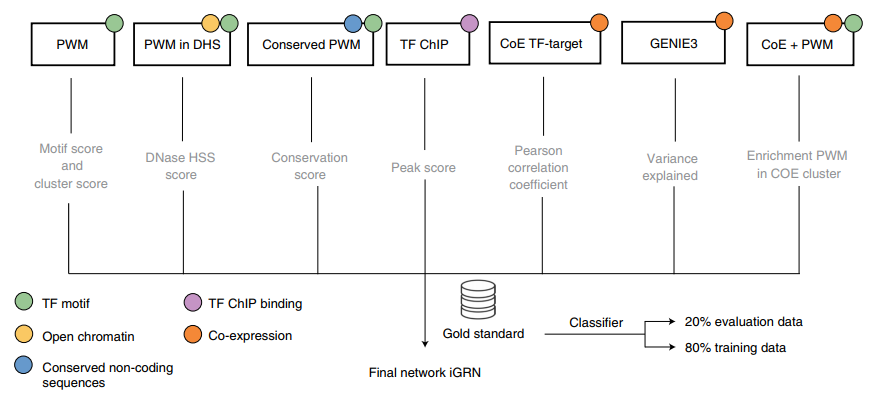
作为基因调控网络,发现新功能、挖掘新基因自然是iGRN不可或缺的功能。研究人员根据iGRN准确地推测出了681个转录因子的一个或多个功能,并对数百个转录因子的未知功能进行了预测。为了验证这个预测的准确性,研究人员通过功能缺失突变体或者基因过表达材料等的研究,对iGRN预测的参与氧化胁迫(ROS stress)途径的转录因子进行检测,成功鉴定出了13个从未被报道参与ROS途径的转录因子,再次说明了了iGRN的可靠性。
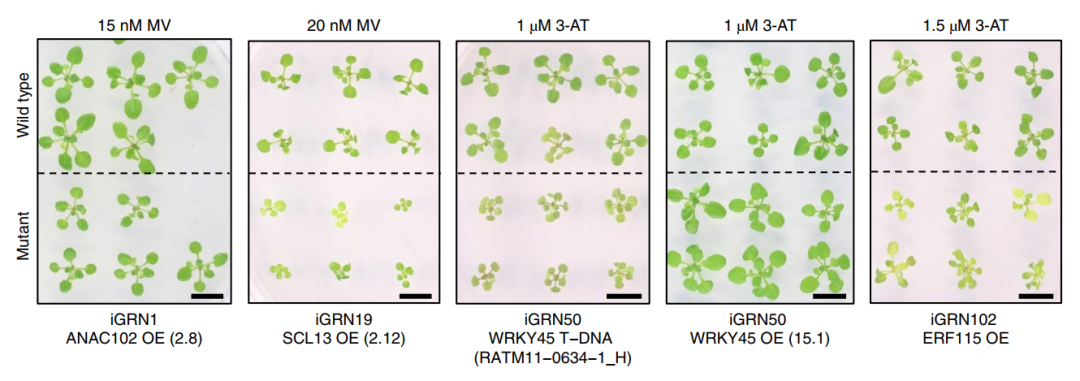
预测的未知ROS基因表型鉴定
综上所述,iGRN是一个蛋白与DNA功能性互作的高质量网络,为进一步研究相关基因的调控机制提供了一些新的思路,也为植物整体的基因调控网络提供了一个新的视角。
参考文献:
【1】Franco-Zorrilla, J. M. & Solano, R. Identification of plant transcription factor target sequences. Biochim. Biophys. Acta 1860, 21–30 (2017).
【2】Jones, D. M. & Vandepoele, K. Identification and evolution of gene regulatory networks: insights from comparative studies in plants. Curr.
Opin. Plant Biol. 54, 42–48 (2020).
【3】Gaudinier, A. & Brady, S. M. Mapping transcriptional networks in plants: data-driven discovery of novel biological mechanisms. Annu Rev. Plant Biol. 67, 575–594 (2016).
iGRN:
论文链接: